Genetische Redundanz
Der folgende Artikel gibt den im „Studium Integrale Journal“ (29. Jahrgang ┃ Heft 2; Oktober 2022) erschienenen Artikel „Über den Entwurf des Lebens: Genetische Redundanz“ von Dr. Peter Borger wieder. Zitate ohne weitere Quellenangabe stammen direkt aus dem erwähnten Originalartikel.
Genetische Redundanz könnte für den Laien mit „Fehlertoleranz“ oder „Back up“ übersetzt werden. Es bedeutet, dass Gene nicht egoistisch sind (jedes nur für seinen eigenen Aufgabenbereich zuständig: bestimmte Proteine herzustellen), sondern dass sie in Netzwerken zusammenarbeiten. Das führt dazu, dass eine Aufgabe von mehreren Genen ausgeführt werden kann, zur Ausführung aber nur eins dieser Gene benötigt wird. Wenn also eines dieser Gene ausfällt, übernimmt ein anderes Gen die Aufgabe, sodass es zu keiner Änderung im Phänotyp (Summe aller Merkmale) kommt.
Bei Labormäusen wurde das mit sogenannten „Knockouts“ herausgefunden. „Knockout“ bedeutet, dass ein Gen vollständig ausgeschaltet wird, wodurch das entsprechende Protein nicht mehr gebildet wird. Die Wissenschaftler iniziierten also solche Knockouts in der Erwartung, dass es zu fehlerhaften oder mangelhaften Merkmalsausprägungen im Phänotyp käme. Daraus könnte dann auf die Funktion des Gens geschlossen werden. Heute gibt es für fast alle menschlichen Gene, die auch in Mäusen vorkommen, einen Maus-Knockout.
Die genaue Erklärung, wie diese Knockouts verursacht und bei den Nachkommen erkannt werden, erspare ich dem Laien. Wen es interessiert, kann aber gerne im Kommentar nachfragen. Jedenfalls gibt es einen vollständigen Knockout erst ab der zweiten Tochtergeneration, wenn eine Reinerbigkeit hergestellt wurde, da bekannterweise jeder Organismus zwei Gene von der selben Art hat. – Je eins von jedem Elternteil. (siehe mendelsche Vererbungsregeln)
Überraschenderweise führten die Knockouts aber zu keiner auffälligen phänotypischen Änderung. Im Rahmen einer evolutiven Entstehung sollte jedes einzelne Gen eine messbare Fitness und einen selektierbaren Nutzen haben. Dementsprechend müsste auch jeder Knockout zu einem messbar veränderten Phänotyp führen. Viele Knockouts haben aber keine phänotypische Änderung zur Folge oder erst nach der Inaktivierung weiterer Gene.
Das bedeutet, dass es Gene gibt, die verzichtbar sind. Sie können ausgeschaltet werden, ohne dass es – oder nur sehr geringe – Auswirkungen auf den Phänotyp hat. Viele Gene machen sogar den Anschein keine einzige messbare Funktion zu haben (Trotzdem können sie eine unbekannte Funktion haben).
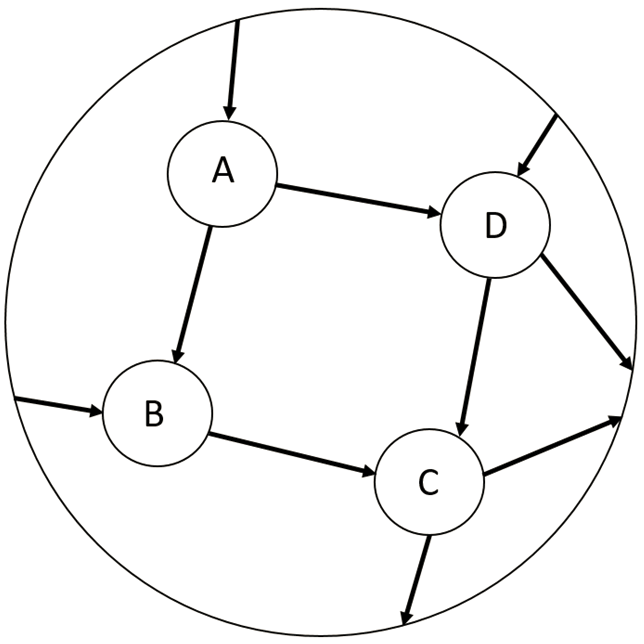
Diese Grafik veranschaulicht das Prinzip der genetischen Redundanz:
Zu sehen ist ein Ausschnitt von 4 Knoten eines komplexen biologischen Netzwerkes. A kann C über B oder D erreichen. Wenn bspw. B ausgeschaltet wird, kann C immer noch über D erreicht werden, wodurch es zu keiner phänotypischen Änderung kommt.
ggggggggggggggggggggggggggggggggggggggg
ggg
Durch verschiedene Knockout-Experimente und Untersuchungen von natürlich vorkommenden Knockouts wurde gezeigt, dass genetische Redundanz die Regel ist, nicht die Ausnahme. Damit kommen wir zum Problem für die Evolutionstheorie:
In dem Buch „Evolution by Gene Duplication“ von Susumo Ohno wird die Idee behandelt, dass genetische Redundanz durch Genduplikation entstanden ist. Manchmal wird ein Gen oder ein längerer Abschnitt biologischer Information während der Zellteilung dupliziert. Findet die Duplikation in Keimbahnzellen statt und wird damit vererbbar, kann das gleiche Gen doppelt in den Nachkommen vorkommen. Ohno schlägt vor, dass Duplikationen genetische Redundanzen schaffen, welche anschließend Mutationen anhäufen und neue biologische Funktionen übernehmen können.
Die Argumentation lautet, dass duplizierte DNA-Elemente nicht der natürlichen Selektion unterliegen und somit frei in neue Gene umgewandelt werden können. Es wird davon ausgegangen, „dass sich ein dupliziertes Gen im Laufe der Zeit aufgrund von Mutationen in den regulatorischen und codierenden Segmenten des Duplikats in seinen Ausprägungen oder seiner Funktion vom Ursprungsgen unterscheiden kann.“ Vorteilhafte Duplikate sollen von der Selektion bevorzugt werden, während sie zeitgleich die Redundanz alter Funktionen schützt, wenn neue Funktionen entstehen.
Nach Ohnos Schätzung sollen für jedes neue duplizierte Gen ungefähr 10 redundante Kopien zu den funktionslosen DNA-Basensequenzen dazukommen müssen.
Diese Idee der Evolution durch Duplikation erklärt auch die Knockouts ohne phänotypische Änderung: Es ist plausibel, dass in gewissem Maß Redundanz zu erwarten ist, wenn Gene sich relativ oft duplizieren, „da Duplikate einen Organismus mit Ersatzgenen versorgen.“ Ohnos Theorie sagt damit einen Zusammenhang zwischen genetischer Redundanz und Genduplikation voraus.
Diese Frage wurde von Biologen an den zahlreichen Daten von der gewöhnlichen Bäckerhefe untersucht. Ganze 60% der Gene der Bäckerhefe konnten inaktiviert werden, ohne dass sich der Phänotyp veränderte. Doch 1999 berichteten Winzler und seine Mitarbeiter in der wissenschaftlichen Fachzeitschrift Sience, dass nur 9% der verzichtbaren Gene von der Bäckerhefe Sequenzähnlichkeiten mit anderen Genen im Hefegenom haben und somit nur diese 9% das Ergebnis von Genduplikation sein könnten.
91% der verzichtbaren (nicht aller Gene!) Gene in der gewöhnlichen Bäckerhefe sind also nicht mit anderen Genen im Hefegenom verwandt (wenn man Ähnlichkeit als Maß der Verwandschaft deutet), was den Schluss nahe legt, dass sie nicht durch Genduplikation entstanden sind.
Das wurde später durch weitere Wissenschaftler bestätigt (Wagner 20001; Kitami & Nadeau 20022). Genetische Redundanz ist nicht das Ergebnis von Genduplikation, sondern von Wechselwirkungen zwischen nicht verwandten Genen. Die Vorhersage aus Ohnos Theorie hat sich also nicht erfüllt und damit hat sich seine Theorie als falsch erwiesen.
Ein weiteres Argument lautet: „Wenn die Evolution von proteincodierenden Genen hauptsächlich auf (nahezu) neutrale Mutationen zurückzuführen wäre, dann sollten Mutationen in Genen, die der reinigenden Selektion weniger ausgesetzt sind, häufiger erhalten bleiben, da sie nicht selektiv ausgemerzt werden. Solche nicht-essenziellen Gene sollten sich leichter und schneller verändern können.“
Diese Vorhersage konnte aber intensiver wissenschaftlicher Forschung zum Trotz nicht bestätigt werden:
- Eine systematische Analyse von Mäusegenen hatte zum Ergebnis, dass sich essenzielle Gene nicht langsamer verändern als verzichtbare Gene.3
- E. coli-Proteine, die in großen redundanten Netzwerken arbeiten, können ebenso viele Mutationen tolerieren wie einzigartige Single-Copy-Proteine.4
- Bei Genomvergleichen von Mensch und Schimpanse stellte sich heraus, dass nicht-funktionale Pseudogene, die als Redundanzen betrachtet werden können, einen ähnlichen Prozentsatz an Nukleotidsubstitutionen haben wie essenzielle proteincodierende Gene.5
Somit ist auch dieses Argument nicht bestätigt.
Gene arbeiten in Netzwerken zusammen, überschneiden sich in Funktionen, können sich gegenseitig ersetzten und übergehen. Einzelne Ausfälle können verkraftet werden, da Gene nicht linear (wie eine Kette) angeordnet sind, sondern als Netz. Und das ist ein Problem für die Evolutionstheorie und gleichzeitig ein Indiz für einen Ingenieur:
Das Vorhandensein von Vorkehrungen für mögliche künftige Ausfälle oder Fehler zeugt von Zukunftsplanung, was typisch für einen intelligenten Ingenieur ist, nicht aber mit reinen Naturprozessen erklärt werden kann. Da redundante Gene nicht ständig gebraucht werden, unterliegen sie nicht der Selektion. Das bedeutet, dass schadhafte Mutationen diese Gene zerstören können, ohne dass die Selektion diesen Prozess verlangsamen könnte. Im Rahmen einer Makroevolution über Millionen Jahre könnten redundante Gene also niemals so lange überlebt haben. Redundante Gene können einige tausend Jahre alt sein, aber niemals Jahrmillionen.
Genetische Redundanz widerspricht der ET und stellt ein klares Schöpfungsindiz dar.
Literatur:
- Wagner A (2000) Robustness against mutations in genetic networks of yeast, Nat. Genet. 24, 355-361. ↩︎
- Kitami T & Nadeau JH (2002) Biochemical networking contributes more of genetic buffering in human and mouse metabolic pathways than does gene duplication. Nat. Genet. 32, 191-194. ↩︎
- Hurst LD & Smith NGC (1999) Do essential genes evolve slowly? Curr. Biol. 9, 747-750. ↩︎
- Hahn MW, Conant GC & Wagner A (2004) Molecular evolution in large genetic networks: does conectivity equal constraint? J. Mol. Biol. 58, 203-211. ↩︎
- Nachman MW & Crowell SL (2000) Estimate of the mutation rate per nucleotide in humans. Genetics 156, 297-304. ↩︎

